标签:har 附件 size 序列 data creat 分享 erp 信息
关于蛋白质对中可能相互作用域的数目的计算这篇论文

CREATE TABLE PFAM(DomainAccchar(7) PRIMARY KEY,DomainId varchar(256),DomainDesc varchar(256),InterproIdchar(10));CREATE TABLE GO(GoTermchar(10) PRIMARY KEY,Ontology varchar(256),GoDesc varchar(256));CREATE TABLE PGMAP(DomainAccchar(7),GoTermchar(10),PRIMARY KEY (DomainAcc,GoTerm),);CREATE TABLE INTERACTION(Domain1char(7),Domain2char(7),iPfam boolean,3didboolean,ME boolean,RCDP boolean,Pvalueboolean,Fusionboolean,DPEA boolean,PE boolean,GPE boolean,DIPD boolean,RDFF boolean,KGIDDI boolean,INSITE boolean,DomainGAboolean,PP boolean,PredictionConfidencechar(2),SameGOboolean,PRIMARY KEY (Domain1,Domain2),);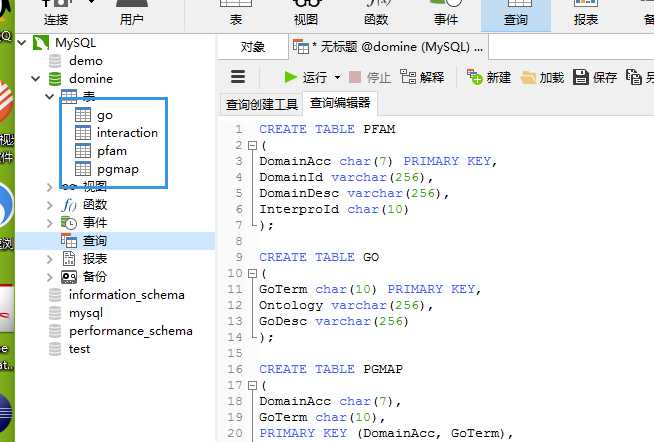

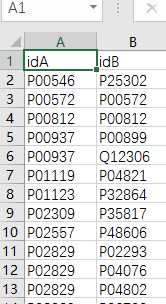
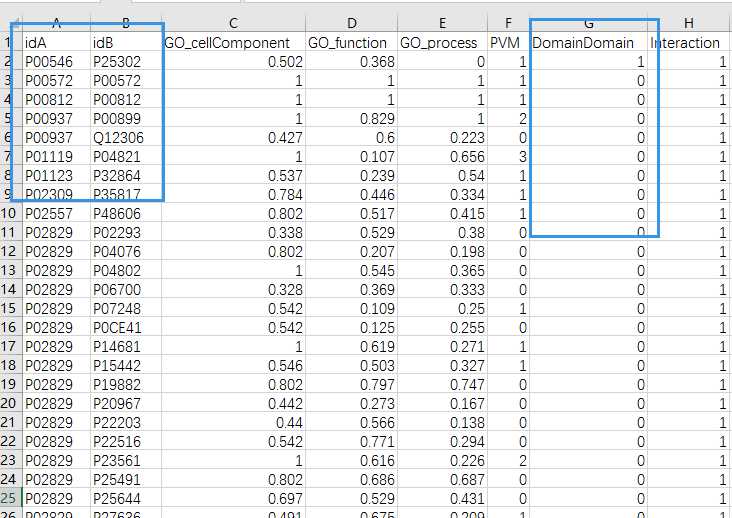
# -*- coding: utf-8 -*-"""Created on Fri Nov 04 15:40:03 2016@author: sun"""importMySQLdbimport pandas as pdimport reyeast_gold_protein_pair=pd.read_csv(‘yeast_gold_protein_pair.csv‘,usecols=[‘idA‘,‘idB‘])yeast=pd.read_csv(‘yeast.csv‘,usecols=[‘Entry‘,‘Gene ontology IDs‘],index_col=0)idA=yeast.loc[yeast_gold_protein_pair.idA,:]idB=yeast.loc[yeast_gold_protein_pair.idB,:]idA.index=range(len(idA))idB.index=range(len(idB))db =MySQLdb.connect("127.0.0.1","root","123","domine")cursor = db.cursor()results=[]for i in range(len(idA)): go_a=tuple(re.findall(r"GO:\d{7}",str(idA.loc[i]))) go_a=‘\‘,\‘‘.join(go_a) go_b=tuple(re.findall(r"GO:\d{7}",str(idB.loc[i]))) go_b=‘\‘,\‘‘.join(go_b) sql_a ="select * from pgmap where goterm in (‘%s‘)"% go_a sql_b ="select * from pgmap where goterm in (‘%s‘)"% go_b# 执行SQL语句 a=cursor.execute(sql_a) results_a = cursor.fetchall() b=cursor.execute(sql_b) results_b = cursor.fetchall()if(len(results_a)!=0and len(results_b)!=0): results_a=tuple(re.findall(r"PF\d{5}",str(results_a))) results_a=‘\‘,\‘‘.join(results_a) results_b=tuple(re.findall(r"PF\d{5}",str(results_b))) results_b=‘\‘,\‘‘.join(results_b) sql="select * from interaction where domain1 in (‘%s‘) and domain2 in (‘%s‘)"%(results_a,results_b) result=cursor.execute(sql) results.append(result)else: results.append(0)yeast_gold_protein_pair[‘domain‘]=resultsyeast_gold_protein_pair.to_csv(‘domain.csv‘,index=False)# 关闭数据库连接db.close()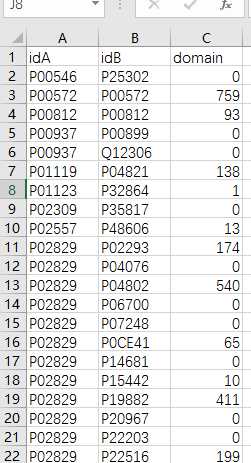
标签:har 附件 size 序列 data creat 分享 erp 信息
原文地址:http://www.cnblogs.com/ahusun/p/6068926.html